先转一下多线程的概念:
线程是一个单一的执行流程,它是所有程序执行过程中最小的控制单位,即能被 CPU 所调度的最小任务单元。线程与进程之间既有联系,又完全不同。简单地说,一个线程必然属于某一个进程,而一个进程包含至少一个或者多个线程。早期的计算机系统一次只能运行一个程序,因此,当有多个程序需要执行的时候,唯一的办法就是让它们排成队,按顺序串行执行。进程的出现打破了这种格局,CPU 资源按时间片被分割开来,分配给不同的进程使用。这样一来,从微观上看进程的执行虽然仍是串行的,但是从宏观上看,不同的程序已经是在并行执行了。如果我们把同样的思想运用到进程上,很自然地就会把进程再细分成更小的执行单位,即线程。由于一个进程又往往需要同时执行多个类似的任务,因此这些被细分的线程之间可以共享相同的代码段,数据段和文件句柄等资源。有了进程,我们可以在一台单 CPU 计算机系统上同时运行 Firefox 和 Microsoft Office Word 等多个程序;有了线程,我们可以使 Firefox 在不同的标签里同时加载多个不同的页面,在 Office Word 里编辑文档的同时进行语法错误检查。因此,线程给我们带来了更高的 CPU 利用率、更快速的程序响应、更经济地资源使用方式和对多 CPU 的体系结构更良好的适应性。关于多线程的详细讲解,可参看:perl 线程模型讲解(http://it.chinawin.net/softwaredev/article-124a1.html)
————————————————————–
perl中的多线程模块
5.8以后的版本的多线程模块可参看perldoc(http://perldoc.perl.org/threads.html)
—————————————————————
perl的多线程实例:
涉及语言:Perl
所用模块:threads
模块中的方法: threads->create(),
创建一个新线程;threads->join(),
收割已经创建的线程;threads->list(threads::all),
返回所有已经创建的线程;threads->is_joinable(),
返回目标线程是否已经完成,等待join;
其他的在perldoc上了。
——————————————————–
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 | |
—————————————————
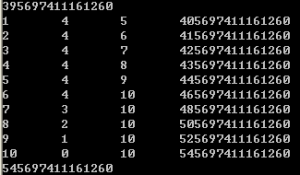
结果:
第一列表示程序已经完成的任务数,第二列表示正在运行的线程数-1(join掉一个了),第三列表示在收割掉一个线程后新添加的任务,最后一列表示完成一个线程时的系统时间。
————————————————————
多线程运行性能
如果单独运行这10个任务,所需要的时间为:1+2+3+4++10=55s;
采用多线程运行(5个)的话,需要的时间为:54-39=16s;
————————————————————-
运行过程
简要描述一下程序运行过程,以便更深入理解多线程的概念。
程序共要运行10个任务,第一个任务的作用是暂停程序1s(sleep(1));第二个任务是暂停程序2s(sleep(2));以此类推,第十个任务是暂停程序10s;
时间(s) 任务
0 1,2,3,4,5(程序初始,5个线程同时运行,需要时间最长的是线程5(5s))
1 2,3,4,5,6(经过1s后,第一个任务已经完成,被join掉,同时添加新任务6)
2 3,4,5,6,7(同上)
3 4,5,6,7,8
4 5,6,7,8,9
5 6,7,8,9,10
7-end join所有剩下的线程(所有任务都已经添加,程序中while循环退出)
方法$thread->is_joinable()的作用
前面已经说了,这个方法是用来判断线程是否已经运行完成,处于等待join的状态。当需要处理多个任务,但这些任务完成需要的时间又不一样时,这个方法就显得特别重要。
还是以上面的程序为例。程序初始运行时创建5个线程。第一个线程所需时间最短,为1s。第五个线程所需时间最长5s。如果不适用$thread->is_joinable()而直接join这五个线程的话,如下:
foreach $thread(threads->list(threads::all))
{ $thread->join();
}
结果是:主程序处于等待状态。在1s后,第一个线程被join,主程序依然处于等待,2s后第二个线程被join,主程序等待。知道5s后第五个线程被join,主程序通畅,重新创建下一组线程(5个)。显然这个过程不能最大话利用CPU的资源。当第一个线程被join后,虽然程序中只有4个线程在运行,但是由于主程序处于等待状态,新的线程不会被创建。
最佳的方法就是判断线程是否可以被join。如上面的程序所写的。这样可以保证程序运行过程中始终是5个线程,最大化的利用CPU资源。
——————————————————-
实例
说了这么多,多线程在生物信息中到底可以怎么来运用,下面给一个简单的实例。从KEGG数据库(http://www.genome.jp/kegg/)上搜索同源序列。
所需文件:seqname.txt(用于存放需要搜索的序列KEGG名称);
源码:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 | |
————————————————